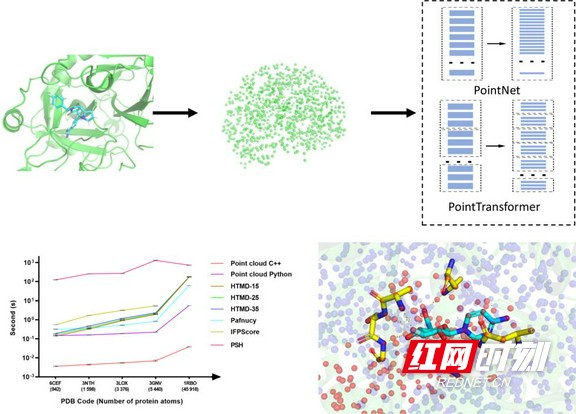
基于点云的蛋白-配体亲和力预测,左下图为处理速度比较,右下为预测结果可视化。
红网时刻11月29日讯(通讯员 刘祥)近日,中南大学湘雅国际转化医学联合研究院段燕文团队在国际生物信息学权威期刊《Briefings in Bioinformatics》(JCR一区, IF 11.622)在线发表了题为“A point cloud-based deep learning strategy for protein-ligand binding affinity prediction”(DOI:10.1093/bib/bbab474)的最新研究成果。2019级硕士生王叶继为论文的第一作者,通讯作者段燕文和黄勇,该研究提出了一种点云深度学习算法实现蛋白-配体亲和力预测的策略。
点云(point cloud)是某一坐标系下点的集合,可以包括点的三维坐标及自身属性。蛋白-配体亲和力预测是药物设计的重要手段,点云用于蛋白-配体亲和力预测尚未见报道。该研究利用基于点云的深度学习算法,将PDBbind数据库中的蛋白-配体结构信息转化为11327个点云后,分别使用PointNet和PointTransformer模型进行训练,预测蛋白-配体亲和力。与多个最新报道的方法相比,这一新的策略在数据前处理速度上具有较大的优势,该方法同样可提取蛋白和配体的相互作用特征,并可以将特征描述符应用于机器学习。最后,基于点云的深度学习方便实现在原子水平的可视化,有助于理解算法的学习过程。
β-Rubromycin(β-RUB)是一类具有人类端粒酶抑制活性的高度氧化芳香族聚酮化合物,具有针对新靶点的抗肿瘤新药开发前景,但由于其溶解度和产量极低等原因限制了其进一步的应用开发。通过特异性探针,从GenBank数据库中高效挖掘出RUB类生物合成基因簇,并成功地发现了β-RUB的天然高产链霉菌CB00271,并通过发酵优化将其中β-RUB 产量提高了12.6倍(超过到120 mg/L),是目前报道的β-RUB最高产量。在此基础上,还分离发现了一个新的β-RUB类似物β-RUB酸,表现出更好地血清溶解性和抗肿瘤活性。该成果以“Genome mining of Streptomyces sp. CB00271 as a natural high-producer of β-rubromycin and the resulting discovery of β-rubromycin acid”(DOI:10.1002/bit.27732)为题,发表在《Biotechnology & Bioengineering》(JCR一区),2017级博士生易理伟为论文的第一作者,通讯作者段燕文和朱湘成。
Rubiginones属于芳香族聚酮的angucycline家族,它们能够增强长春新碱诱导的针对其耐药癌细胞系的细胞毒性。团队通过对链霉菌CB02414的基因组挖掘,发现了两个II型聚酮的生物合成基因簇。其中一个负责孢子色素的生物合成,另外一个则可以合成角环蒽醌类天然产物,其中包含两个细胞色素P450编码基因。通过对这两个生物合成基因簇中关键基因的敲除和过表达,发现了一类新的rubiginone类天然产物;通过对积累的化合物的结构分析,推测了rubiginone类天然产物的生物合成途径,特别是rubiginone类化合物A环上的氧化修饰过程。该成果以“Genome mining of novel rubiginones from Streptomyces sp. CB02414 and characterization of the post-PKS modification steps in rubiginone biosynthesis”(DOI: 10.1186/s12934-021-01681-5)为题,发表在《Microbial Cell Factories》(JCR一区),2018级硕士生张景琰为论文的第一作者,通讯作者段燕文教授和颜晓晖研究员。
中南大学湘雅国际转化医学联合研究院上述工作得到了教育部111项目、国家自然科学基金、中南大学研究生创新基金和中南大学高性能计算平台的支持。
来源:红网
作者:刘祥
编辑:易锐华
本文为健康频道原创文章,转载请附上原文出处链接和本声明。
本文链接:https://health.rednet.cn/content/2021/11/29/10471242.html
 时刻新闻
时刻新闻